科维创生物科技
KeScience
联系电话
400-0660-885
CRISPR操作指南-敲低(敲除)人源circRNA1635
一、背景(介绍circRNA产生的一般原则和Alu元件) 二、如何操作(如何精确找到circRNA两侧毗邻的Alu元件-两个实例) 三、题外话(circRNA过表达成环载体介绍及效率比较) 一、背景
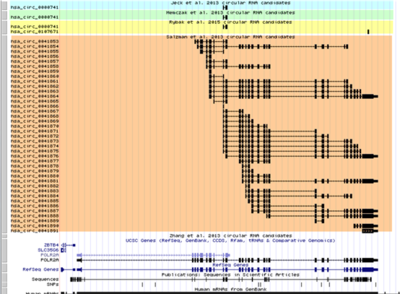
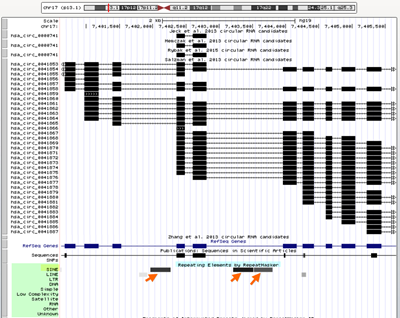
circbase数据库里POLR2A基因位产生的circRNAs示意图。 不同的颜色highlight的是不同研究组报道的circRNAs,其中最上面3个研究组都发现了一个共同的circRNA-circPOLR2A,下图5讨论的也正是这条circRNA。 通常情况下circRNA的产生遵循以下原则(图1和2):
环状RNA 的基因组特征 1 中间的外显子最容易成环,通常包含2~3个外显子;如果是一个外显子成环,则其长度会明显长于平均长度; 2 成环外显子的两侧内含子较大(~5倍于随机内含子); 3 通常,circRNA两侧毗邻内含子含有互补的序列,如短间隔重复序列(SINE)家族(图4)的Alu元件(the inverted repeated Alu elements,IRAlus)等,且距离成环外显子边界距离相当(图2和3)。
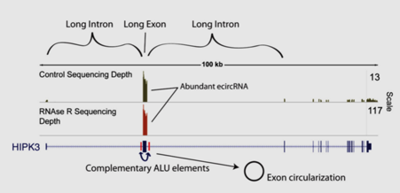
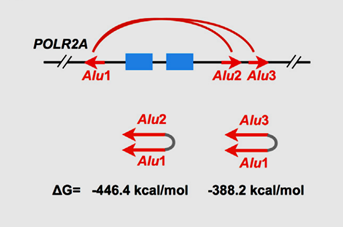
POLR2A基因位产生的其中一个circRNA-circPOLR2A及其毗邻的IRAlus示意图。 Zhang, et al. (2014). "Complementarysequence-mediated exon circularization." Cell159(1):134-147. Alu元件是灵长类基因组中一组散在分布的相关序列,每个长约300bp。单个Alu元件成员的每个末端上有Alu限制酶的切割位点,并由此命名。在灵长类的基因组中存在着大量不同种类的Alu元件。事实上,Alu元件是人类基因组中丰度最高的转座元件。特别地,Alu元件在人的基因组中占比11%,在影响基因表达等方面影响广泛。Alu元件基本组成模式如下: PartA--AAAAATACAAAAAA--Part B-polyA,其中A和B序列往往类似。 但通常成环外显子两侧毗邻的内含子较大,其中包含的重复序列,如Alu元件等也往往是多个并存(图4)。简单的原则是:距离最近的IRAlus优先考虑。但实际的情形具体如何、能否高效去除内源circRNA的形成等问题还需要具体问题具体分析。
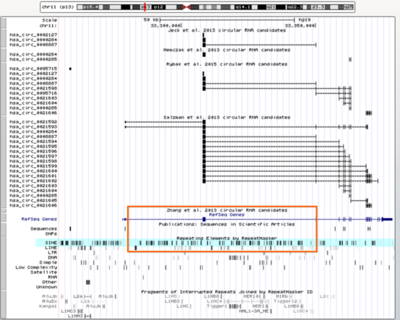
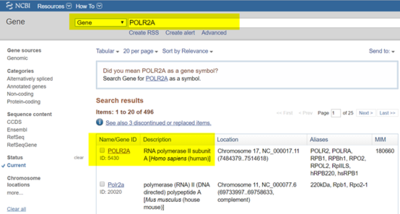
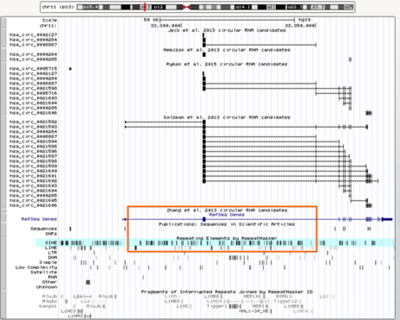
circHIPK3的基因组特征—两侧毗邻内含子中的重复序列(SINE)。 里面包含几十个SINE元件,但具体哪个(哪些)介导了成环,需要谨慎的下结论(来源于circbase)。 二 如何操作 在本文中我们以map circRNA的IRAlus为例,示例如何找到元件的边界,从而利于下游的CRISPR操作(CRISPR操作可以参考实验小白公众号系列文章-shiyanxiaobai): 已知:circRNA的ID、序列 目的:Mapping到两侧毗邻内含子中IRAlu(反向配对)序列 工具:Snapgene、RepeatMasker、NCBI blast l circPOLR2A的例子 1) circBase调取hsa_circ_0000741序列 可通过circBase调取,方法请参考“circRNA引物设计教程”。 2) 下载人POLR2A基因的genebank格式文件(.gb格式) 登陆NCBI-gene下搜索POLR2A(图5),找到物种人的序列(第一条就是)点击进去,拉到genebank(图6),进入(图7),按照顺序选择并保存文件。
3) 然后文件用snagnene打开即可。
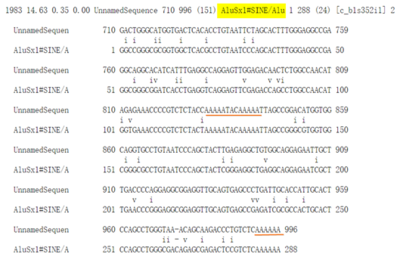
Snapgene文件打开.gb格式的genebank文件。外显子/内含子组织情况非常清晰。 下面的circRNA和Alu序列是确定序列之后手动注册上去的。 4) 根据circbase调取的序列把circRNA对应到环化的外显子上(图8蓝色的两个线条对应的两个外显子)。 5) 然后分析两侧毗邻的内含子序列,看看是否含有重复序列如IRAlus等。其实circbase里面提供了这一数据,如图9。数据显示两侧内含子共包含3个SINE元件---AluSz、AluY、AluSx1.
数据库显示成环相关的2个内含子含有3个SINE元件,其中左边1个,右边2个(来源于circbase) 6) 然后分析左侧的AluSz和右边的AluY、AluSx1能否组成IRAlu对即可。首先通过RepeatMasker网站(http://repeatmasker.org/cgi-bin/WEBRepeatMasker)找到AluSz、AluY、AluSx1的序列(图10-12)。
AluSz序列。根据Alu元件的结构,明显这是反向的。
AluY序列。根据Alu元件的结构,明显这是正向的。
AluSx1序列。根据Alu元件的结构,明显这是正向的
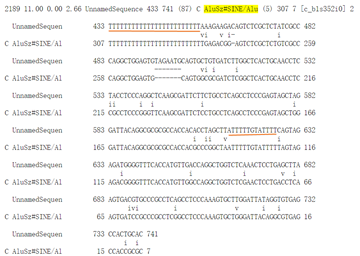
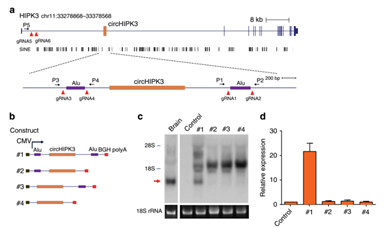
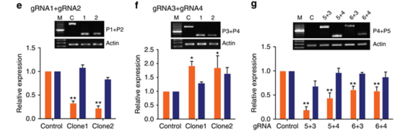
Alu元件Blast比对示意图 8) 设计两条sgRNA对单侧IRAlus做切除即可(CRISPR怎么做?请参考链接系列教程)。 以上过程非常愉悦,问题不大。但现实里往往有让人崩溃的例子。 l circHIPK3的例子 circHIPK3(重复使用图4,见下)的成环相关的左侧内含子包含28个SINE,右侧内含子含有51个SINE。但具体是哪个反向互补的SINE元件介导成环根本不清楚。有研究报道,环化外显子两侧的IRAlus一般距离剪切位点在~1kb左右。 所以,2016年的NCpaper的做法就是分别克隆成环外显子上下游各~1k的距离在体外test成环效率(~1kb范围内只有一对IRAlus),发现删除任何一个Alu都影响体外成环(图14);然后作者就利用CRISPR的手段删除基因组上的对应的Alu并test成环效率。最终发现情况比较复杂,其中删除右侧的Alu效果相对最好(图15,grna1+2)。这间接反映出IRAlus之间可能存在复杂的相互作用,也说明circRNA的产生是一个极其复杂、调控精巧的过程。 (开脑洞:如果把两侧内含子中的主要元件都做切除结果会如何?再开脑洞:如果过表达IRAlus的RNA或者DNA互补序列能否干扰circRNA的形成?能否优于siRNA/shRNA的敲减?)(这篇文章做的是比较常规的细胞功能实验,机制也是经典ceRNA。即使如此,也发了10分以上的文章)
circHIPK3的基因组特征—两侧毗邻内含子中的重复序列(SINE)。 里面包含几十个SINE元件,但具体哪个(哪些)介导了成环,需要谨慎的下结论(来源于circbase)
circHIPK3示意图。体外实验发现删除两侧任何一个Alu都会极大降低成环效率。 Zheng, Q., et al. (2016). "Circular RNA profiling reveals anabundant circHIPK3 that regulates cell growth by sponging multiplemiRNAs." Nat Commun7: 11215.
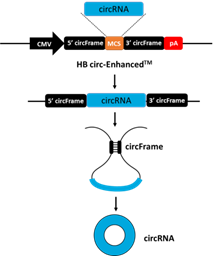
接上图,利用CRISPR技术删除circHIPK3两侧Alu元件对成环的影响。 Zheng,Q., et al. (2016). "Circular RNA profiling reveals an abundant circHIPK3that regulates cell growth by sponging multiple miRNAs." Nat Commun7: 11215. l 总结: Map circRNA成环的SINE如IRAlus元件是一件相对复杂和繁琐的工作,过程也充满了诸多不确定性。因此,这是一件探索性很强的实验,而且结合大热门的CRISPR基因组编辑技术,可以极大提高文章的质量,希望能力较强的同学多多尝试。 一、题外话: 在诸多test之后,科维创生物独家研发了一款高效成环过表达载体系统---pHBcirc-EnhancedTM(图16),这一高效成环系统可以保证克隆的circRNA 100%成环(实例见下),这一系统通常包装成病毒出售。除此之外,考虑到一部分的人不会操作病毒,我们也同时推出了非病毒载体的成环系统构建试剂盒—pHBcirc-BasicTM。这一基础款的成环载体基本可以满足前期circRNA的筛选验证工作。
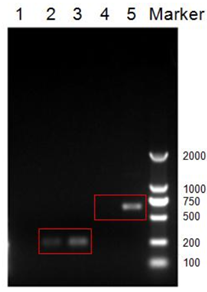
科维创环化载体高级版pHBcirc-EnhancedTM示意图。 上下游的成环元件是经过精心设计、优化和验证过的,可以达到100%成环的效果。 科维创生物pHBcirc-EnhancedTM成环效率对比实例: 1. 根据“circRNA引物设计教程”从circbase调取环状RNA的序列,模板DNA合成并进行载体构建; 2. 用LipoFiterTM转染试剂转染 293T 细胞,48 hr后提取总 RNA,使用Oligo dT和随机6碱基引物进行反转录; 3. 根据“circRNA引物设计教程”设计引物,其中mmu_circ_0000880 PCR产物预期~200 bp,mmu_circ_0000199 PCR产物预期~700 bp; 4. 用Agrose胶进行分析,结果如图17; PCR产物测序确认成环接口序列。
1. 空载对照 2. 某赛克隆载体-mmu_circ_0000880 3. pHBcirc-EnhancedTM-mmu_circ_0000880 4. 某赛克隆载体- mmu_circ_0000199 5. pHBcirc-EnhancedTM-mmu_circ_0000199 |